本篇目录:
ChIP-seq数据质控与过滤
1、大部分的ChIP-seq数据都是短读长,去低质量不是必须的。但是fastqc图中大量碱基质量如果存在明显的下降,那么就需要去掉 常规的去除方法是 :对每条read,从检测到Q值低于某个阈值(比如Q20)开始去除,直到恢复正常的Q值。
2、ChIP-seq测序后该做哪些生物信息学分析 高通量数据类型主要包括基因芯片和基因测序,我估计你想知道的是具体的内容。
3、分析ATAC-Seq从本质上来看和分析ChIP-Seq没啥区别,都是peak-calling,也就是从比对得到BAM文件中找出reads覆盖区,也就是那个峰。那么问题集中在如何找到peak。
4、ChIP-Seq的原理是:首先通过染色质免疫共沉淀技术(ChIP)特异性地富集目的蛋白结合的DNA片段,并对其进行纯化与文库构建;然后对富集得到的DNA片段进行高通量测序。
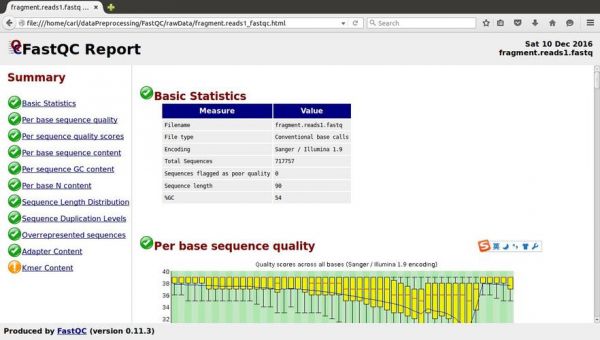
5、新手,刚做完一个ChIP-Seq项目的分析,来记录一下,会分好几篇。首先是下机数据fastqc之后会生成一个html格式的报告,根据报告可以看出自己数据的特点,便于之后clean的参数设置。
FastQC的基本介绍
1、FastQC是一款基于Java的软件,须在linux环境下使用命令行运行,它可以快速多线程地对测序数据进行质量评估(Quality Control),其官网地址为: Babraham Bioinformatics 。FastQC可以使用conda进行安装。
2、Multiqc能将fastqc生成的多个报告整合成一个报告(HTML和PDF格式),方便的查看所有测序数据的质量。Multiqc支持多种分析类型的质控结果查看,包括:RNAseq、Whole-Genome Seq、Bisulfite Seq、Hi-C等。
3、fastqc函数,输入样品fq格式文件,输出样品的zip压缩包。
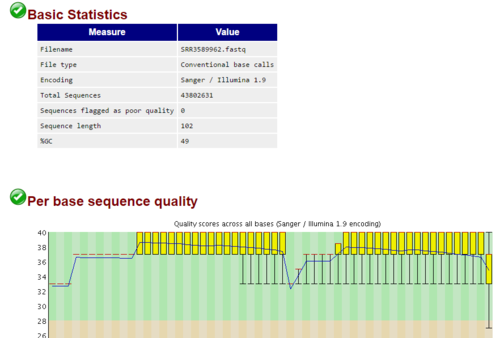
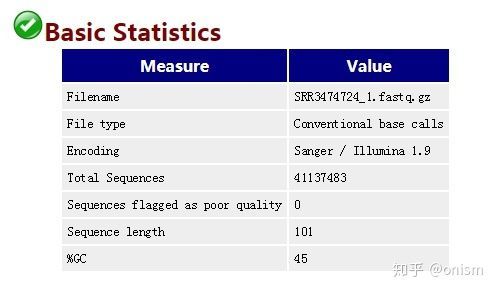
4、每个fastqc文件会获得一个质量分析报告,来描述此次RNA-seq的测序质量。 获取质量报告如图:从read水平来总览,判断测序质量。 Encoding :测序平台的版本,因为不同版本的 error p的计算方法不一样。
5、在测序实验之后,包括RRBS或WGBS,需要对数据进行预处理。
6、此步骤的可用软件:CASAVA和FastQC 测序运行完成后,在开始分析之前,应检查运行的质量是否以下参数,这些参数可能说明样品和运行的质量。
二代测序为什么要用到linux
1、有以下几个原因: 软件开发:Linux是开源的,程序员可以看到和修改源代码,并且大多数服务器都运行在Linux上。学习Linux可以帮助软件开发人员了解操作系统底层的实现机制,提高开发效率和优化代码。
2、二代测序又称为高通量测序(High-throughput sequencing),是基于PCR和基因芯片发展而来的DNA测序技术。关于二代测序的临床应用:二代测序作为一种检测手段,主要应用于基因的生殖变异(遗传性)与体细胞变异(获得性)的检测。
3、应用高通量技术进行转录组测序是一种快捷可靠的获取转录组信息的方法。mRNA的转录本表达分析,通过获得研究对象基因组转录区域的信息,鉴定转录发生位点,可变剪切等,其精确的计数方法更可对基因进行精确的定量分析。
4、我们都知道一代测序为合成终止测序,而二代测序开创性的引入了可逆终止末端,从而实现边合成边测序(Sequencing by Synthesis)。
5、因为在IT领域,linux占据很重要的地位,你将来从事的工作,都要使用到linux,例如: 改天你做开发,程式码要放到伺服器,伺服器基本上都用linux,那么你要会操作。
6、rTn5转座酶的插入位点具有很高的随机性,因此被广泛的用于体外转基因(外源基因整合到宿主细胞)和二代测序建库等领域。
2022你还在用fastqc?超高速fastq前处理工具教程
1、方法/步骤进入NCBI主页,选择Nucleotide数据库 在Nucleotide数据库的检索框中输入甘油醛-3-磷酸脱氢酶基因的基因名(GAPDH)或者基因的GenBank号:X02661。点击搜索。
2、使用命令 fastqc -o seqfile1,seqfile. 来进行质量报告。需要注意的是./fastqc前面的.不可以省略 每个fastqc文件会获得一个质量分析报告,来描述此次RNA-seq的测序质量。
3、fastp不仅可以自动识别fastq数据里的引物,匹配序列,还能自动识别数据是single end还是pair end支持长/短read序列。常用测序平台的引物和匹配序列fastp都会自动识别不需要手动指定。并且还能自动识别读序错误进行删除。
FastQC质控报告解读
一般FastQC分析结果产生有2种类型的文件,一种.zip,一种.html。直接点击.html文件,即可在浏览器中查看质控结果报告。首先是Summary,这一部分是整个报告的目录,整个报告分成若干个部分。
首先是下机数据fastqc之后会生成一个html格式的报告,根据报告可以看出自己数据的特点,便于之后clean的参数设置。
使用命令 fastqc -o seqfile1,seqfile. 来进行质量报告。需要注意的是./fastqc前面的.不可以省略 每个fastqc文件会获得一个质量分析报告,来描述此次RNA-seq的测序质量。
到此,以上就是小编对于的问题就介绍到这了,希望介绍的几点解答对大家有用,有任何问题和不懂的,欢迎各位老师在评论区讨论,给我留言。

 微信扫一扫打赏
微信扫一扫打赏